I ricercatori hanno utilizzato il principio dell'evoluzione per scoprire rapidamente le molecole più efficaci contro le malattie.
Come funziona un farmaco? Agisce sulla proteina bersaglio coinvolta in una malattia e la disinnesca. Così come gli anticorpi naturali nel nostro corpo.
Ma come si trova la molecola che avrà il giusto effetto su questa proteina? Si usa una tecnica per setacciare le molecole ad alto rendimento per vedere quale funzionerebbe meglio.
Lo screening tradizionale ad alta produttività viene eseguito in piastre da 96 o 384 pozzetti e ogni molecola viene testata in un singolo pozzetto.
Molto laboriosa, questa tecnologia, automatizzata (con un robot per erogare liquidi, movimentare i piatti, ecc.).
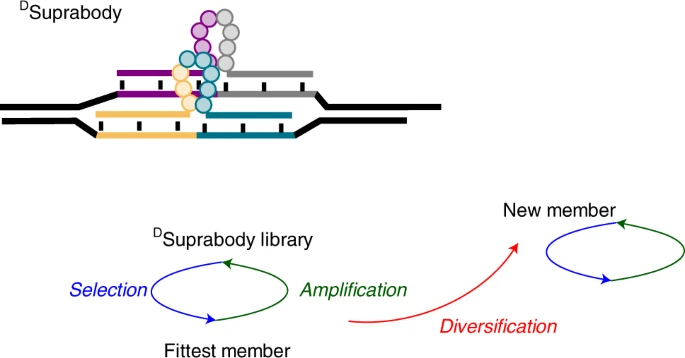
È stata migliorata alcuni anni fa sulla base del DNA di queste molecole, semplificando, così, la loro identificazione. Ma gli scienziati dell'Università di Ginevra hanno appena scoperto di meglio: una tecnica che impiega solo una o due settimane per identificare la migliore combinazione possibile, una tecnica ispirata a... Charles Darwin.
Il naturalista britannico ha infatti dimostrato che l'evoluzione naturale favorisce i migliori individui mentre genera diversità per meglio adattarsi al cambiamento.
Lo stesso hanno fatto i chimici dell'UNIGE: 'La biologia trova sempre una soluzione a un problema', spiega Nicolas Winssinger, professore presso il Dipartimento di Chimica Organica della Facoltà di Scienze dell'UNIGE e autore della corrispondenza dello studio pubblicata questo mese. il 6 dicembre in 'Natura'.
'È questo principio di evoluzione naturale che abbiamo messo in atto per le piccole molecole'.
Hanno così sviluppato una tecnologia che genera diversità creando più di 100 milioni di assemblaggi di molecole attraverso il loro DNA, che poi selezionano per abbinare al meglio una particolare proteina.
Concretamente, qui, lo screening viene effettuato immobilizzando la proteina bersaglio su piccole sfere di resina. “La collezione di molecole (100 milioni) viene aggiunta e quelle che hanno le migliori affinità rimangono attaccate dalle loro interazioni con la proteina bersaglio.
Poiché ogni molecola della nostra collezione è etichettata con una sequenza di DNA e il DNA è facilmente amplificabile (questa è chiamata reazione a catena della polimerasi, o PCR, la stessa tecnologia utilizzata per rilevare SARS-CoV2 in un test: una molecola di DNA è sufficiente), noi possiamo analizzare le molecole selezionate. In altre parole, molecole che interagiscono con la proteina bersaglio, o molecole di interesse.
Quando inizi con collezioni fino a 100 milioni, ci saranno molti 'vincitori', come fai a sapere quale molecola è davvero la migliore? Il tag DNA ci permette anche di ricomporre il vincitore.
In altre parole, puoi amplificare il risultato di una prima selezione e ripetere la selezione una seconda volta e così via”.
Tutto questo avviene in una o due settimane, rispetto a mesi o addirittura un anno per gli schermi a banda larga tradizionali.
I ricercatori di Ginevra hanno testato il loro metodo con la proteina PD-L1. Ciò impedisce al sistema immunitario di attaccare le cellule cancerose spacciandole come parte del corpo umano.
'Grazie alla nostra metodologia, abbiamo rapidamente identificato un assemblaggio che mira specificamente e disinnesca il PD-L1, confermando che funziona in modo efficace', afferma Nicolas Winssinger.
Inoltre, questa tecnica è facile da riprodurre in qualsiasi laboratorio del mondo e costa solo poche migliaia di euro, rispetto ai milioni per lo screening ad alta produttività.
'Sfruttando le forze dell'evoluzione evidenziate da Darwin, possiamo ora migliorare i nostri assemblaggi di molecole e aprire un nuovo spazio di possibili combinazioni che non è stato ancora sfruttato, al fine di creare nuovi farmaci più efficienti', conclude il ricercatore.
Nessun commento:
Posta un commento